It компании помогают геномной медицине
Московский центр исследований и разработок EMC по облачным вычислениям и большим данным в Сколково работает над проектом создания новой технологии для поиска информации в хранилищах данных со сложной структурой связей между элементами. В таких системах требуется обход графа* для нахождения подходящих элементов и определения их похожести.
Технология будет необходима везде, где нужно работать c большими коллекциями данных различной природы – прежде всего на уровне метаданных (содержательных описаний массивов информации), позволяя находить и использовать заранее не очевидные связи между объектами.
Одним из важнейших и актуальных направлений применения системы является геномная медицина, где объемы данных велики, и имеют колоссальное число внутренних и внешних связей (ссылки на публикации, на близкие термины в онтологиях и т.п.)
В своей финальной стадии он направлен на создание удобной инфраструктуры, которая поможет врачу и исследователю найти потенциально ценную для лечения информацию гораздо эффективнее и интеллектуальнее, чем это происходит сейчас.
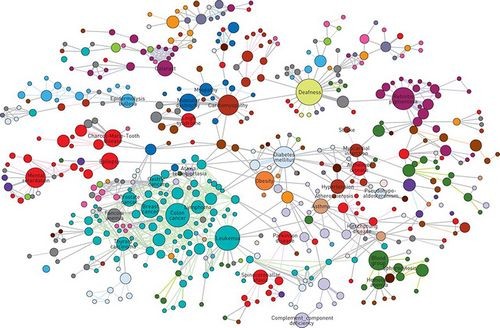 Пример того, как выглядит граф
Пример того, как выглядит граф
Источник: Goh K et al. «The Human Disease Network», PNAS 2007; 104: 8685-8690Проблематика
Существует множество онтологий – сложно структурированных «словарей», ссылающихся друг на друга. К примеру, есть онтологии болезней, генов, базы ассоциаций (установленных связей между, например, генетическими вариантами и вероятностью тех или иных заболеваний), и т.д.
Еще есть «факторы окружения»: к ним относятся курение, употребление алкоголя, радиация при лечении рака, лекарственные препараты – всё, что влияет на вероятность возникновения болезни, её течение и лечение. Для их описания тоже используют онтологии – например, онтологию лекарств DrugBank, онтологию биологически значимых химических соединений CHEBI, объединённую онтологию фармацевтических веществ RxNorm и т.д.
Для описания фенотипа, в данном случае диагнозов заболеваний, при создании системы использовались онтологии DOID (или просто DO – Disease Ontology), а также HPO (Human Phenotype Ontology), для описания генотипа – GO и faldo. Сейчас система развивается, для описания диагнозов добавляется ICD10, для описания генетических аспектов – RefSeq и GenCode.
В совокупности это терабайты данных. Чтобы ориентироваться в этом потоке информации, нужно правильно сформировать запрос и настроить поиск.
Но именно здесь и есть основное препятствие: данные имеют разную структуру, формат, содержат множество ссылок друг на друга и на внешние источники. Это колоссальные объемы литературы и в открытом, и закрытом доступе.
Массивы могут быть разбросаны по Интернету или не выходить за пределы одной клиники. Информация часто не формализована, не стандартизирована.
Возникает вопрос, как корректно и эффективно делать запросы одновременно к многочисленным рассеянным базам данных, имеющим разную структуру, и получать осмысленный ответ о связях различных составляющих находящейся там информации (геномных, фенотипических, факторов окружения и тд.)? Самое важное – как находить и учитывать не только явно заданные ссылками связи между элементами данных, но и смысловые, неявные связи.
Решение проблемы
Московский центр исследований и разработок EMC видит решение проблемы в создании системы на основе проиндексированной многофункциональной базы данных. Концепция в том, чтобы объединять массивы данных из самых разных источников, анализировать их и выявлять связи.
Пилотная версия системы позволяет специалисту отправить запрос ко всему массиву, найти данные за доли секунды и получить развернутый ответ с учетом всех возможных связей, и что важно – контекста, даже если прямого указания на него нет. Так, врач может посмотреть, например, какие лекарства использовались при конкретных или сходных болезнях при разных вариантах геномных параметров, и получить ответ не только на свой прямой вопрос, но и на близкие вопросы, которые могут ему помочь в принятии решения.
Проект сейчас находится на исследовательском уровне, впоследствии его использование в медицине и науках о жизни будет также обсуждаться в рабочих группах Глобального альянса по геномике и здравоохранению (GA4GH). В полном объеме аналогов этой технологии пока нет.
Работа системы
Сначала система кластеризует, то есть объединяет большие данные по схожести (даже если прямой связи нет) и фильтрует их. В случае большой выборки первой появляется та информация, которая лучше всего отвечает на запрос поиска.
Для этого используются различные механизмы ранжирования обнаруженных результатов по их релевантности.
Бывают ситуации, когда искомой информации мало и необходимо использовать связи с другими фрагментами данных. Разрабатываемый Центром EMC проект помогает находить данные вместе с облаком контекста, в котором они находятся.
Пользователь может задать конкретное лекарство, болезнь и ген. В ответ система выдает неочевидную, на первый взгляд, связь этих параметров.
Она может подсказать специалисту пути к более эффективному лечению.
Таким образом, система станет важным и ускоряющим компонентом для ежедневной работы специалиста, которому сейчас даже в самых продвинутых клиниках мира приходится самостоятельно, руками, при отсутствии единого интерфейса искать информацию по многочисленным базам данных.
Важно отметить, что система не является рекомендательным сервисом: программа ничего не советует, а ускоряет поиск необходимых данных и автоматизирует целый ряд процессов. Решения по лечению и другим целям принимаются самими специалистами.
Реализация концепции предполагает, что поисковый «движок» можно будет развёртывать на площадях клиентов. Это позволяет включить в поиск данные по пациентам госпиталей.
Обычно такие данные запрещается передавать третьим лицам, а организовать поиск по таким базам при помощи публичных сервисов зачастую невозможно.
Можно будет использовать федеративный поиск – нахождение информации на удаленных источниках. В функционале есть возможность отключать выдачу конкретного типа данных, включать поиск по публичным и собственным базам.
Система поможет медицинским специалистам самого разного уровня:
• она ускорит работу врачей, которые до сих пор самостоятельно ищут информацию в многочисленных источниках.
• она создаст нужную инфраструктуру для клиник. Система архивирует информацию в многофункциональной базе данных, в которой сохраняются и сами документы, и связи между ними.
В результате у клиник будет возможность собрать накопленные знания, удобно укомплектовать и эффективно в них ориентироваться, пользуясь лишь ноутбуком.
Проект, над которым работает команда российских специалистов вместе с иностранными коллегами из компании EMC, уже получил положительные отзывы на выставке-конференции в области информационных технологий, биоинформатики и геномной медицины BioIT World, прошедшей в начале апреля 2016 года. Параллельно налаживается сотрудничество с крупной американской клиникой, которое позволит проверить работу системы в приложении к клинической практике.
В российских реалиях эта система также может быть применена, и Московский центр исследований и разработок EMC по облачным вычислениям и большим данным открыт к сотрудничеству.
Вышеописанный функционал – лишь часть пилотного проекта. В финальной версии система уже сможет анализировать целые группы пациентов с различными параметрами генов, симптомов, заболеваний.
Что особенно важно, геномными данными возможности технологии не ограничиваются. В дизайне системы нет ничего сугубо специфического для медицины и наук о жизни, поэтому область ее применения в будущем можно сильно расширить.
* Здесь граф – это структура данных, нечто вроде социальной сети разного рода описаний, документов и т.п., где в узлах сети документы, а связи между ними указывают, как один документ связан с другим. Например, определение руки содержит ссылку на описание пальцев.
Определение рака ссылается на разные виды рака, они – на разные подвиды. Одна болезнь может быть связана с другой не только как подвид, но и как родственная структура.
Леонид Левкович-Маслюк, директор по науке и инновациям в RD Центре EMC?
Как попасть в топ IT компании